- 🔗 Conexión
- 🗂️ Transferencia de archivos
- 📄 Edición de archivos
- 📦 Crear un ambiente con las dependencias necesarias
- 🖥️ Correr código
- 🛠️ Cosas útiles
- Redirigir la salida de la consola a un archivo
- Gestión de experimentos
- Correr scripts de Python con parámetros
- Obligar a Jax a usar la CPU incluso si la GPU está disponible
- Encontrar bottlenecks en el código
Desde el subsistema Linux, podemos conectarnos al cluster usando ssh.
ssh user@cluster.ing.uc.cl
donde user es el nombre de usuario. Va a pedir una contraseña.
La forma más cómoda en mi opinión de traspasar tu código al cluster es creando un repositorio con tus archivos, subiéndolo a GitHub, y luego clonando ese repositorio desde el cluster.
Hay varios comandos que te conecten con el repositorio remoto, como:
- Descargar el código con
git clone(si es el repositorio es privado) - Obtener los cambios que se hayan hecho en el repositorio desde la página de GitHub, con
git pull - Subir a GitHub el código que editaste en la copia del repositorio en el cluster, con
git push
Cada vez que necesites conectarte al repositorio remoto tendrás que autentificarte con tu usuario y tu contraseña. Si esto se vuelve muy tedioso en algún momento, es posible hacer una "llave" con la que acceder, ver Generating a new SSH key and adding it to the ssh-agent - GitHub Docs para más detalles.
Eventualmente podrías necesitar acceder desde el cluster a un archivo que no puedes descargar, o descargar archivos desde el cluster a tu computador. Una opción para hacer esto es un SSH File Transfer Protocol.
Desde el subsistema Linux en tu computador, puedes conectarte al cluster con el comando sftp, que te permite ver tus archivos locales y los archivos remotos simultáneamente, y transferirlos de un lado a otro.
Para conectarse, simplemente usar
sftp user@cluster.ing.uc.cl
Una vez conectado al cluster, puedes revisar tus archivos como lo harías normalmente con ls, moverte de directorio con cd o ver tu directorio actual con pwd. Además de eso, puedes revisar tus archivos locales con lls, moverte localmente con lcd, o ver tu directorio local con lpwd.
Para descargar un archivo file.txt desde el cluster, te mueves en el cluster al directorio donde está el archivo usando cd, te mueves en tu computador al directorio de descarga con lcd, y descargas el archivo con get file.txt. Similarmente, puedes subir archivos con put.
Para más detalles del uso puedes usar help, que da una lista de todos los comandos disponibles.
Otra opción es sincronizar carpetas; a veces uso esto para transferir archivos desde la carpeta de resultados del cluster a una carpeta local donde puedo trabajar y hacer gráficos. Para esto uso el comando rsync, que permite que el directorio DEST tenga cada uno de los archivos del direcotio SRC. Su sintaxis es
rsync [optional modifiers] [SRC] [DEST]
Un ejemplo de uso:
rsync -rva user@cluster.ing.uc.cl:ACIP-MRI/results/pseudo_golden_hyper_optim results/pseudo_golden_hyper_optim
Correr este comando localmente permite que el directorio local results/pseudo_golden_hyper_optim (es necesario que existe previamente) contenga todo lo que está dentro de la carpeta ACIP-MRI/results/pseudo_golden_hyper_optim de mi cuenta en el cluster. Algunas detalles interesantes:
v(por verboso) hace que se listen en consola los archivos sincronizados.r (por _recursivo_) permite copiar un directorio y sus subdirectiorios también. Sin esta opción, al intentar sincronizar una carpeta con subdirectorios, se obtiene un avisoskipping directory`.a(por archivo) copia no solo el contenido de los archivos, sino también los permisos y fechas.
💡 Tip Para verificar que los archivos que se van a traspasar son los correctos, la opción
-vse puede combinar con la opción--dry-run, que corre el comando sin hacer ninguna modificación en los archivos.
Recursos:
Si necesitas hacer cambios pequeños en los archivos, el cluster tiene instalado el editor de texto vim. Vim suele ser un editor difícil de usar si no estás acostumbrado. Puedes acceder a un tutorial bastante bueno con el comando vimtutor (se ejecuta desde afuera, no desde vim).
A modo de mini tutorial:
- Se puede abrir un archivo en Vim usado
vim file.txt. - Vim tiene varios modos: los más comunes son el modo normal, que está por defecto cuando entras a Vim, y el modo insertar, que deja borrar y agregar texto (también te deja usar
Ctrl + v). Puedes entrar al modo insertar desde el modo normal apretando la letrai. Puedes volver al modo normal con la teclaEsc. - La forma común de trabajar en Vim es mantenerte en modo normal, moverte por el archivo usando las teclas
h,j,k,lhasta llegar a un punto que te interesa editar, entrar al modo insertar usandoi, hacer un cambio pequeño, y luego salir con Esc. - Puedes deshacer cambios con
u. OJO, todo lo que haces desde que entras al modo insertar hasta que sales cuenta como UN SOLO CAMBIO. Por eso conviene solo hacer cambios pequeños, para no perder grandes trozos de tu trabajo. - Para guardar los resultados, aprietas
:wqdesde el modo normal. Para salir sin guardar, aprietas:q!.
Hay muchos atajos de teclado para moverse en el texto, copiarlo, cambiarlo, etc. Ver vimtutor para más detalles.
Recursos:
Yo prefiero trabajar desde un editor de texto más cómodo, como VSCode. Permite conectarse con SSH a algún servidor remoto, y es mucho más cómodo editar los archivos.
⚠️ Atención Descubrí que VSCode tiene un problema: la sesión nunca termina de correr en el cluster, y no he encontrado forma de reiniciarla. Esto es problemático si pides que te instalen cosas después de haber comenzado a usar VSCode, como la sesión nunca termina, las terminales que abres desde ahí nunca encuentran los programas nuevos.
Recursos:
- 🧑🏽🏫 Remote SSH Configuration in VScode
- 📓 Integrated Terminal in Visual Studio Code - Run Selected Text (Una función útil es poder correr el código seleccionado en la terminal).
Para instalar las dependencias, yo uso Conda (puedes pedir que te lo instalen si no lo tienes).
Una complicación con la que me he topado más de una vez es con la instalación de los paquetes para poder usar GPUs. En el cluster hay varias versiones de CUDA instaladas. Se pueden consultar con el comando module av cuda. Yo uso la 11.2 con JAX y no he tenido problemas hasta ahora.
Por defecto no tenemos una GPU, a menos que la pidamos, y eso da problemas en la instalación, porque Conda intenta instalar cosas pensando que no tenemos GPU. Es necesario "engañar" a Conda, usando la variable CONDA_OVERRIDE_CUDA. Ver Tips & tricks — conda-forge 2023.01.11 documentation para más detalles.
A modo de ejemplo, así es como yo hago el ambiente en el que corro el código:
CONDA_OVERRIDE_CUDA="11.2" conda create --name jaxcuda jax cuda-nvcc -c conda-forge -c nvidia
conda activate jaxcuda
CONDA_OVERRIDE_CUDA="11.2" conda install -c conda-forge matplotlib tqdm
CONDA_OVERRIDE_CUDA="11.2" conda install -c anaconda scikit-image ipykernel numpy scipy
pip install wonderwords
Que hace lo siguiente:
- La primera línea crea un ambiente
jaxcudae instala JAX (jax) y CUDA (cuda-nvcc). El orden de los canalesconda-forgeynvidiadesde donde se desgargan los paquetes importa (ver recurso). Aún no tengo claro si es necesario instalar CUDA, puesto que ya está instalada en el cluster. - Activo el ambiente
- Instalo paquetes del canal
conda-forgecomomatplotlib. Notar que cada vez que instalo algo nuevo debo volver a sobreescribir la variableCONDA_OVERRIDE_CUDA. - Instalo paquetes del canal
anaconda. Creo que separar por canales no era necesario, supongo que unconda install matplotlib numpypor ejemplo habría funcionado igual. - Instalaciones con
pip. Recordar instalar conpipdentro del ambiente y solo después de haber instalado todos los paquetes posibles conconda.
⚠️ Atención En mis ambientes instaloipykernelpara poder usarlos con Jupyter Notebooks. Ver más detalles en la sección Jupyter Notebooks, dentro de 🖥️ Correr código
Recursos:
- 📓 google/jax: Composable transformations of Python+NumPy programs: differentiate, vectorize, JIT to GPU/TPU, and more (github.com) - Conda installation
- 📓 Managing environments — conda 22.11.1.post27+ad20af3f6 documentation
Al conectarse al cluster, por defecto estamos en un nodo donde podemos ejecutar código (podemos activar un ambiente Conda por ejemplo y usar Python). Lo ideal es trabajar aquí para verificar que el código esté correcto antes de enviarlo a la cola de trabajos. Aquí no hay una GPU disponible, deben pedirse.
Para enviar a correr un trabajo hacemos un archivo .sh con todas las instrucciones necesarias. Podemos enviar ese trabajo al cluster haciando sbatch run.sh. Usualmente pruebo que todo funcione corriendo el código directamente, sin enviarlo a un nodo de cálculo (con muy pocas iteraciones para que termine pronto) con bash run.sh.
El archivo .sh debe ser algo así:
#!/bin/bash
# Nombre del trabajo
#SBATCH --job-name=NombreTrabajo
# Archivo de salida
#SBATCH --output=salida.txt
# Cola de trabajo
#SBATCH --partition=gpus
# Solicitud de gpus
#SBATCH --gres=gpu:quadro_rtx_8000:1
# Reporte por correo
#SBATCH --mail-type=ALL
#SBATCH --mail-user=usuario@ing.puc.cl
module load cuda/11.2
eval "$(conda shell.bash hook)"
conda activate namecondaenv
python calculo.py
El preámbulo tiene una serie de comentarios SBATCH que dan instrucciones al administrador de trabajos acerca de cómo se va a correr el trabajo. En este caso, estamos pidiendo una GPU quadro_rtx_8000 para correr el trabajo NombreTrabajo. Los outputs que suelen imprimirse en la consola al correr un trabajo serán guardados en un archivo salida.txt. Se nos enviará un correo a usuario@ing.puc.cl notificándonos cuando el trabajo empiece a correr y cuando termine.
La línea module load cuda/11.2 carga CUDA para poder usar la GPU. Luego activo un ambiente namecondaenv y finalmente ejecuto el código python calculo.py.
Recursos:
📓 Envío de trabajos al Clúster – Dirección Económica y de Gestión (uc.cl)
Es posible trabajar de forma interactiva en una GPU (sujeto a la disponibilidad).
srun --job-name "InteractiveJob" --gres=gpu:quadro_rtx_8000:1 --time 24:00:00 --partition=gpus --pty bash
Esto creará un nuevo trabajo InteractiveJob usando una de las GPUs disponibles.
Para poder usar la GPU y que las librerías la detecten, hay que cargar CUDA. Esto se hace con
module load cuda/11.2
Con respecto a la instalación, para trabajar con Jupyter Notebooks y potencialmente varios ambientes de Conda, yo prefiero instalar Jupyper en el ambiente (base), e instalar ipykernel en cada uno de los ambientes donde me interesa usar Jupyter Notebooks, como se explica aquí:
📒 How to set up Anaconda and Jupyter Notebook the right way | by Justin Güse | Towards Data Science
Para que eso me funcione me sirvió esta respuesta.
Pero también se puede instalar Jupyter solo en el ambiente que nos interesa usar.
Para correr un notebook desde el cluster vamos al ambiente donde está instalado Jupyter y usamos el comando.
jupyter notebook --no-browser --port=9876
El puerto port va a ser importante, puesto que el notebook está corriendo en el cluster y a priori no podemos verlo desde el navegador como nos gustaría.
Abrimos una nueva terminal con el subsistema Linux, y sin entrar al cluster escribimos el comando
ssh -t -t tcatalan@mazinger.ing.puc.cl -L 9876:localhost:9876
Poniendo atención de siempre usar el mismo puerto.
Si queremos trabajar con una GPU debemos pedirla primero (ver Uso interactivo de la GPU) y cargar CUDA antes de hacer el notebook. Para poder ver el notebook usamos en lugar de lo anterior:
ssh -t -t tcatalan@mazinger.ing.puc.cl -L 9876:localhost:9876 ssh n7 -L 9876:localhost:9876
De vez en cuando me aparece un error al intentar conectarme mientras uso la GPU
bind: Cannot assign requested address
Que se soluciona cambiando el protocolo a IPv4 según una entrada en un blog (SSH port forwarding: bind: Cannot assign requested address | Electricmonk.nl weblog) que encontré, lo que se traduce en agregar un -4 al comando anterior.
ssh -t -t tcatalan@mazinger.ing.puc.cl -L 9876:localhost:9876 ssh n7 -4 -L 9876:localhost:9876
Si quiero correr un archivo codigo.py, de forma que lo que normalmente imprimiría en consola (prints, errores) se guarde en un archivo de texto, puedo hacer:
python codigo.py &> salida.txt
Para que se imprima al archivo de texto y además se siga viendo en consola
python codigo.py 2>&1 | tee salida.txt
Usualmente corremos muchas versiones de un código cambiando algunos hiperparámetros. Una buena herramienta para gestionar experimentos es Weights & Bias.
- Experimentos reproducibles. No basta con saber qué código se corrió; los hiperparámetros, las librerías usadas y el hardware también pueden alterar los resultados. W&B guarda los detalles necesarios para reproducir el código.
- Evolución del entrenamiento en tiempo real. W&B permite enviar a un servidor distintos objetos que se generan al entrenar un modelo, como métricas, valor de la función de pérdida, imágenes que evolucionan con el entrenamiento (además de videos, tablas y otras cosas). Estos resultados pueden verse desde la página web en tiempo real.
- Framework-agnóstico: se puede usar con cualquier código, no necesita una librería específica. Tiene integraciones que facilitan trabajar con las librerías más conocidas como PyTorch o TensorFlow.
Recursos:
- 🧑🏽🏫 Welcome to Weights & Biases - Introduction Walkthrough (2020)
- 📓 Experiment Tracking: Track and visualize experiments in real time, compare baselines, and iterate quickly on ML projects
Al correr trabajos en el cluster, es conveniente poder modificar los parámetros desde la consola, sin tener que modificar el archivo de Python. Una forma de lograr esto es mediante la librería argparse — Parser for command-line options, arguments and sub-commands. Esta librería permite correr scripts de la forma
python script.py --arg1 1 --arg2 "P1"
python script.py --arg1 2 --arg2 "P2"
El archivo script.py tiene un parser que procesa los distintos argumentos y los entrega en un diccionario.
parser = argparse.ArgumentParser(...)
parser.add_argument('--arg1', type = int)
parser.add_argument('--arg2', type = str)
args = parser.parse_args() # esto es un diccionario Basta con importar Jax y cambiar la configuración, como explica este comentario.
import jax
# Global flag to set a specific platform, must be used at startup.
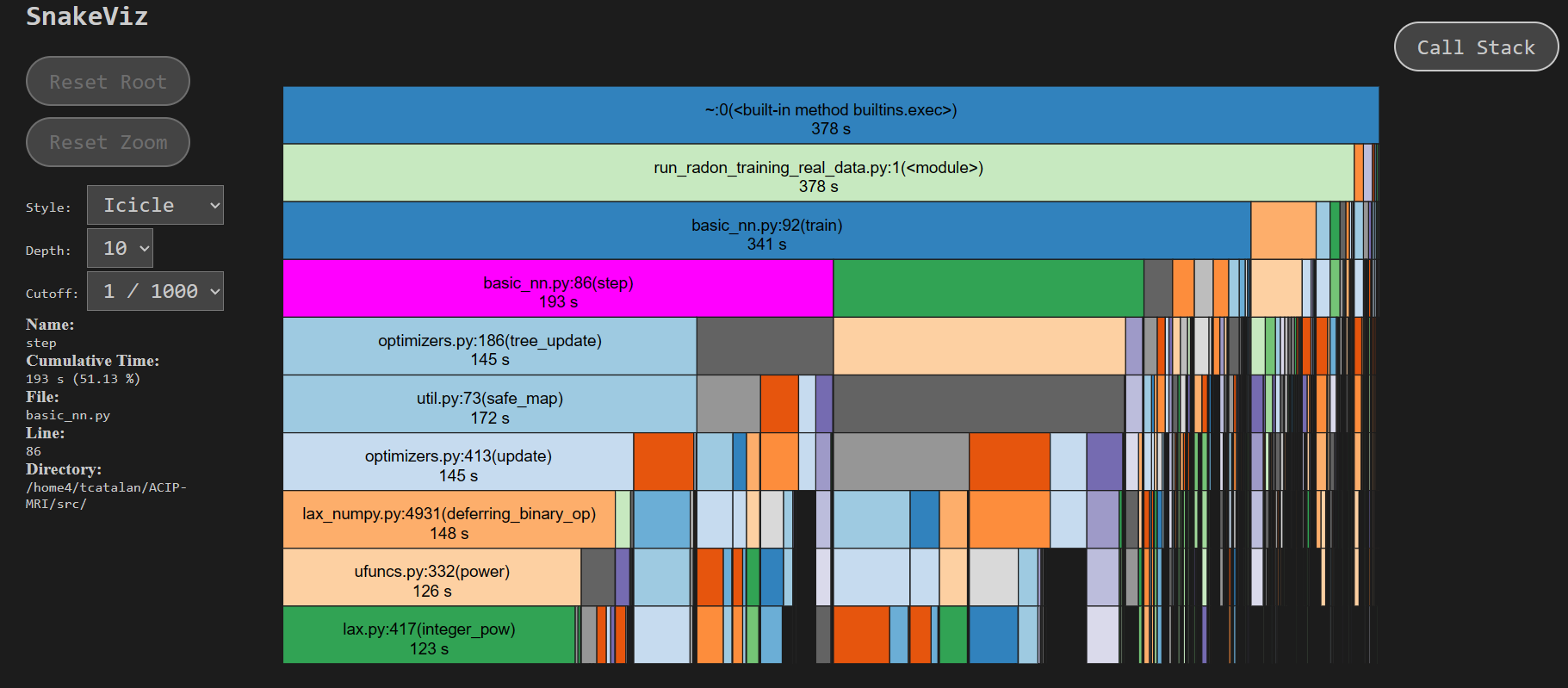
jax.config.update('jax_platform_name', 'cpu')Para estudiar el tiempo de ejecución de un programa se puede usar un profiler, que da una idea de cuánto tarda nuestro programa en cada una de las llamadas que hace. Esto es útil para identificar las funciones que más tiempo toman, o trozos de código que deberían ser optimizados.
Para estudiarlo en programas largos como los entrenamientos de redes neuronales existe cProfile. Hay una alternativa más simple profile pero que hace que el programa tarde más. Para visualizar los resultados puede usarse SnakeViz, que entrega una visualización interactiva así: